基于DNA甲基化特征的乳腺癌异质性分析
看教程不够直观,那就看视频吧! >>点击加载视频
今天给大家分享的内容是“基于DNA甲基化特征的乳腺癌异质性分析”。
在介绍工作内容之前,我们先来了解一些背景知识。
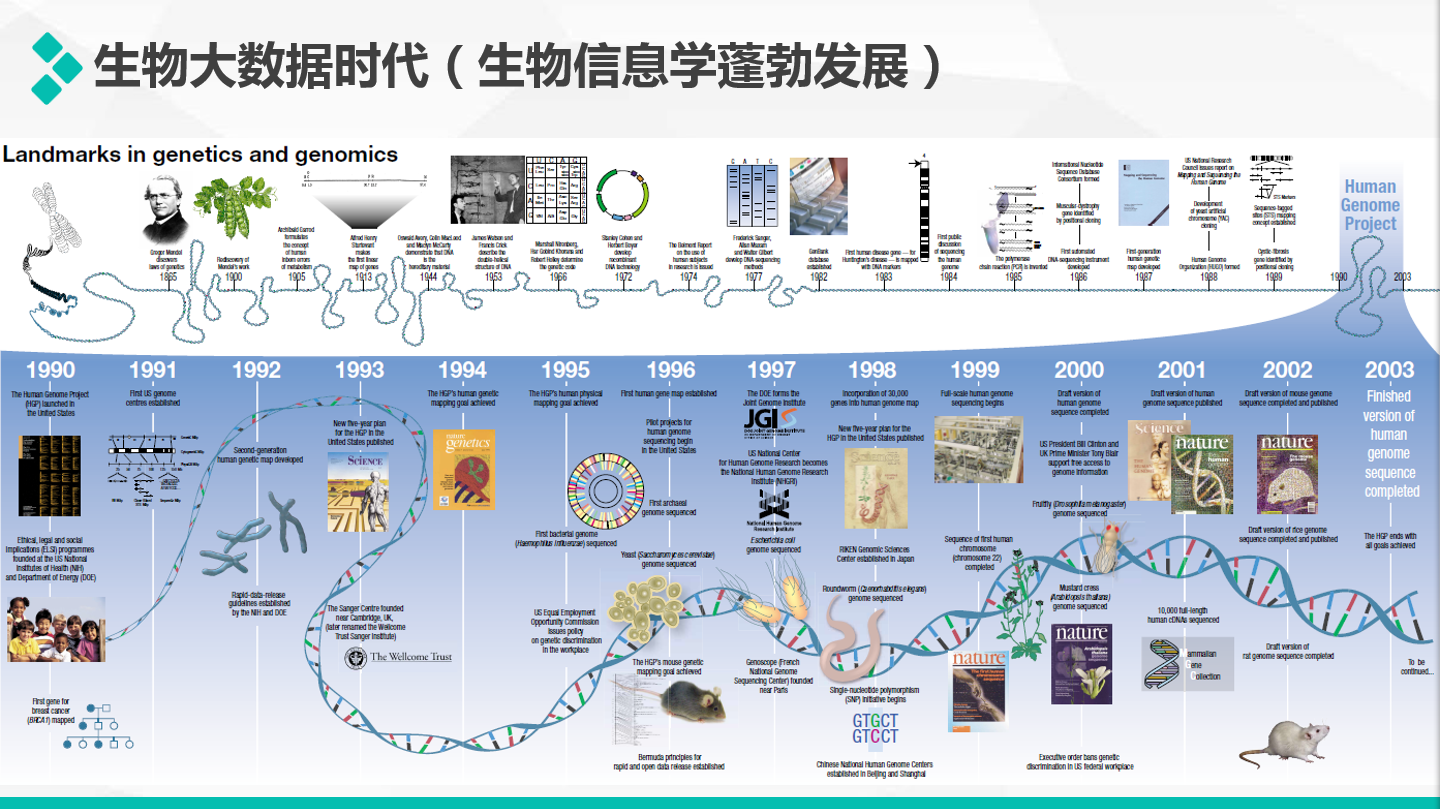

20世纪初期,遗传定律和物种起源的研究成果极大的推动了人们对于生命现象的理解,随着认知的深入和渴望对自身的了解,以及期盼通过分子水平的研究攻克癌症等复杂疾病的诊断治疗,人类基因组计划被启动。随之而来的是测序技术的革新和测序成本的降低,导致生物数据汇聚成浩瀚的海洋,研究人员开始变得一头雾水,为了在大数据中挖掘有用的信息,生物信息数据分析技术应声而出,我们的学院也是这样个时代背景下的产物,她见证了生物数据的井喷,也为生信的发展做出了巨大的贡献。
然而,在实际研究中,人们还是发现有一大部分的生命现象不能被经典的遗传学理论解释。大家本以为“人类基因组”计划完成了,就可以获取病人体内全部的突变位点,进而完成诊断治疗的使命。但却恰恰相反,生命现象的复杂性让科研人员叹为观止。
面对着大量的“DNA序列没有发生改变,但表型却发生了可遗传改变”的实例,人们归纳总结出了表观遗传学的概念:
它是指不需要核苷酸序列变异的基因表达的可遗传改变。

后基因组时代中,人们为了研究更为复杂的基因转录调控等功能基因组内容,于2003年启动了人类表观基因组计划,同时也推动相关领域的快速发展。
那么,表观遗传学究竟是研究什么的呢?在八年制《生物信息学》规划教材中做出了描述,其研究的内容包括两个方面:
第一,转录调控层面,研究DNA甲基化、组蛋白修饰、染色质结构等对转录表达的影响;
第二,转录后调控,研究非编码RNA、miRNA、circRNA等对mRNA的互作及翻译调控。
实现策略就是,通过应用和开发生物信息学方法来解决生物医学相关的表观遗传问题。这也就是我所在的“计算表观遗传学教研室”所做的研究工作。
大量的研究已经发现,疾病的发生与表观遗传修饰之间存在着密切的关系,据epigenomics杂志中的综述归纳,表观遗传参与疾病发生发展的形式可分为三种情况:
第一,表观遗传可能通过介导遗传或环境风险因素参与疾病途径
第二,表观遗传可以作为遗传或环境风险因子的修饰物
第三,表观遗传提供了一种遗传和环境因素参与疾病过程的合理解释
DNA甲基化作为重要的表观遗传修饰之一,大量的研究报道显示其与癌症患者间治疗反应的差异显著相关,并且有研究表明其与肿瘤的复发及转移密切关联。
我们都知道肿瘤是一个复杂的过程,也正是因为他的复杂性和对人类健康的严重威胁,使其成为了重要的研究对象,同时也促进了精准医学在肿瘤研究中的发展。
肿瘤的一个特点是具有高度的异质性,其中不乏基于DNA甲基化所开展的肿瘤异质性的研究。
肿瘤的异质性可分为三个层面,即肿瘤组织内部的异质性,肿瘤患者间的异质性,以及不同类型肿瘤之间的异质性,而最后一部分被认为是一种pan-cancer层面的分析。
那么,针对瘤内异质性,曾有人设计了如下的实验:
将肿瘤组织中的细胞移植到裸鼠上进行成瘤实验,发现大部分细胞移植给小鼠并不会致瘤,而仅有少量肿瘤细胞移植才会致瘤,于是便提出了“肿瘤/癌干细胞”(CSC)的概念。
所以,我们也基于这种癌组织中混杂着多种类型细胞的思想完成了一项工作,即基于DNA甲基化构建细胞亚克隆反卷积模型评估乳腺癌的瘤内异质性。
工作中主要涉及了39个乳腺癌的细胞系和4个正常乳腺细胞系的450K芯片DNA甲基化数据,62个乳腺癌组织的DNA甲基化数据。
利用39个乳腺癌细胞系、2个正常乳腺细胞系和62个乳腺癌组织的DNA甲基化数据进行甲基化水平的分布分析,并构建系统发生树,发现癌症细胞系与癌症组织之间存在着清晰的界线,并且正常的乳腺细胞系独立于二者之间。
通过单独分析,发现乳腺癌组织与细胞系之间存在着三类系统发生关系,即:
① 乳腺癌组织与癌症细胞系具有共同的进化祖先
② 乳腺癌组织与正常细胞系具有共同的进化祖先
③ 乳腺癌组织与正常和癌症细胞系共同具有相似的进化祖先(并且这种类型的系统发生树占很大比例)
基于系统发生树的观察结果及癌症干细胞起源学说,我们设计了“反卷积”的策略来分析和预测癌症组织中的细胞组份及比例,并进而对肿瘤内的异质性进行定量估计。
为了完成这个模型,我们首先基于DNA甲基化水平,利用混合效应模型,筛选了细胞类型特异的基因。
之后,以筛选得到的细胞类型特异甲基化的基因作为反卷积模型的变量因子,通过二次规划和偏最小二乘拟合的方法构建反卷积模型。并在模拟组织数据和真实细胞系数据中进行模型的性能评估,结果均显示出高可性度。
随后,将训练好的模型应用到基于TCGA的乳腺癌450K芯片DNA甲基化谱一致性聚类识别的人群亚组中,发现9个亚组中乳腺癌组织的细胞组份间存在着显著的差异。
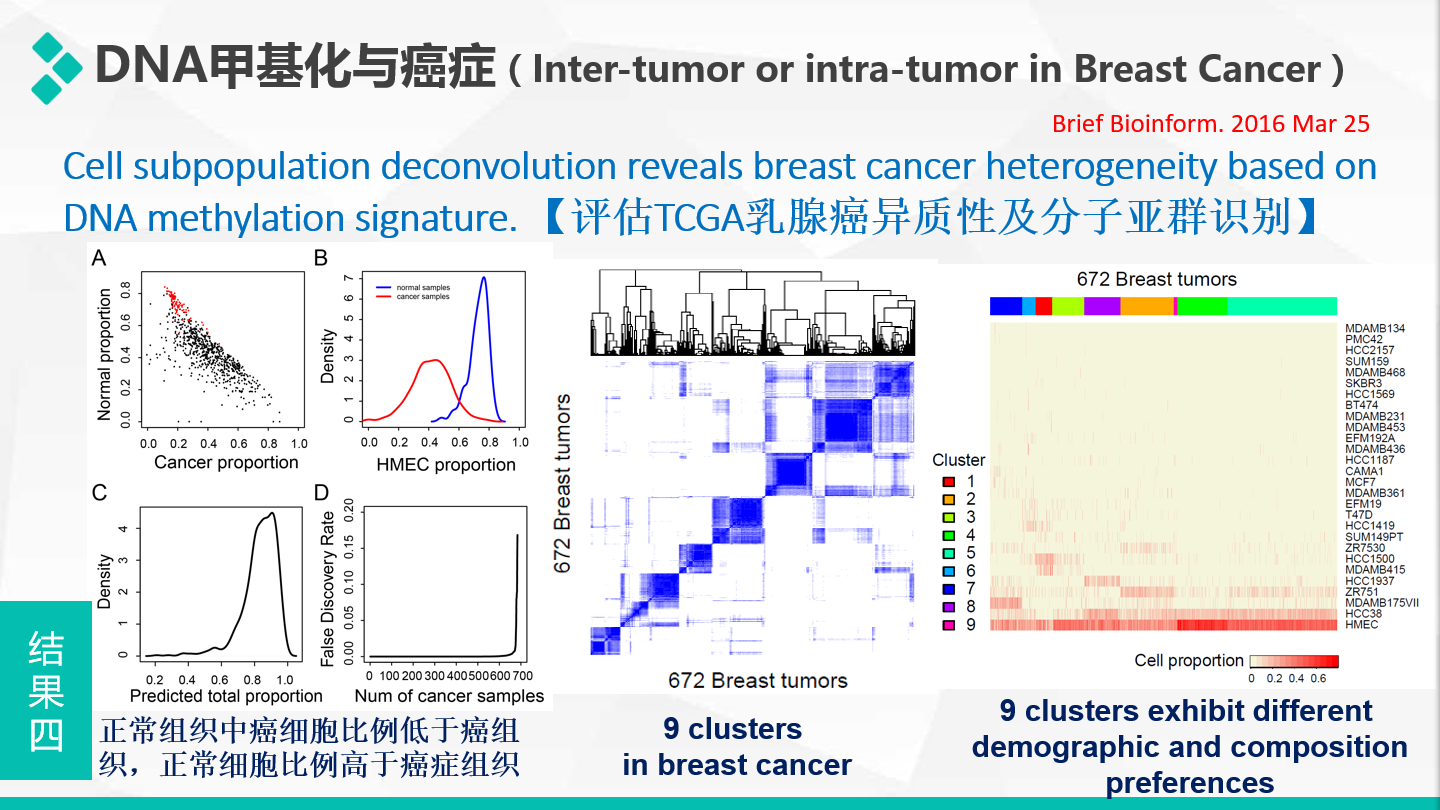
进一步分析,发现9类乳腺癌患者间所富集的临床特征也有显著差异,并且在生存上存在差别。新分子亚型的分类结果中有些高度富集了已知的PAM50、SNF分类信息,这表明细胞类型特异DNA甲基化的基因可用于确定乳腺癌组织中各类细胞亚群的比例,可用于评估乳腺癌的瘤间异质性。
图片编辑起来很是费劲,尴尬,,ԾㅂԾ,,
查看完成版,请移步公众号:
【“生信云”高峰论坛】基于DNA甲基化特征的乳腺癌异质性分析
- 发表于 2017-10-24 09:49
- 阅读 ( 5312 )
- 分类:其他组学
你可能感兴趣的文章
- 生信文章解析(第六篇)揭示MuBC的分子结构 3629 浏览
- 介绍几个DNA甲基化差异筛选的软件 11795 浏览
