Cytoscape——实例
看教程不够直观,那就看视频吧! >>点击加载视频
本文将具体操作怎样用Cytoscape绘制网络图
Cytoscape所支持的数据格式:
1.*.sif格式:
nodeA<interaction>nodeB
nodeC<interaction>nodeD
…
即文件分为三列,第一列和第三列是有相互作用关系的基因名或蛋白质名等,第二列是相互作用的名称
*.sif格式简单,容易处理,但它不能规定每个节点的位置、大小、形状等。
2. xgmml格式,它是一种xml格式,可以规定节点和边的许多信息,但也更复杂。
3.*.txt格式:用tab分割的纯文本文件
可以将文件设置成两列,每一列都是基因名(或蛋白质名),同一行的两个基因(或者蛋白质等)代表有互作关系;也可以加其他参数放在第三列,例如两基因调控的强弱系数
本文以txt格式的数据进行演示绘制网络图
网络文件:net.txt:共表达网络;共四列,前两列是gene id,第三列是共表达类别(正1/负-1),第四列是相关系数,以tab键分隔
节点属性文件:
步骤:
导入网络文件:file->import->network->file(net.txt)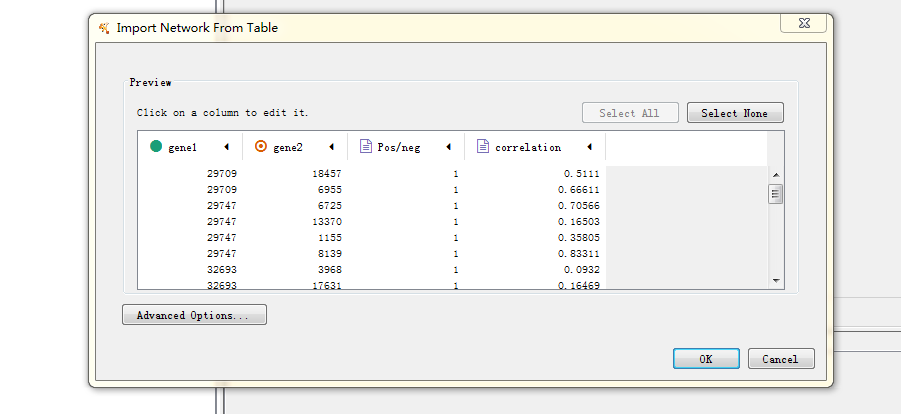
其中不同标识代表着不同的含义
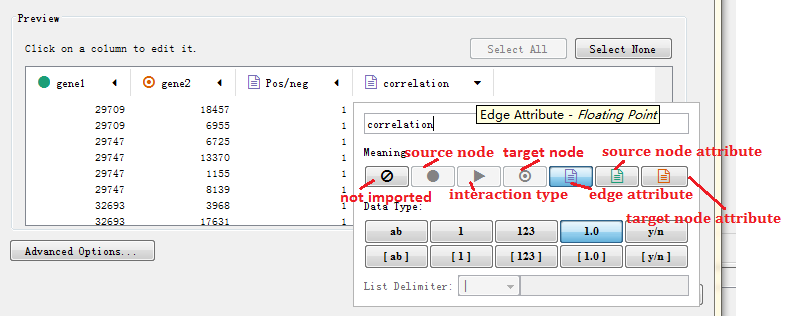
导入后
导入节点属性文件:file->import->table->file(node.txt)(此处为table而非network)
注:node.txt:节点属性文件。四列,包含三种属性;第一列为gene id,与网络文件中一致,第二列为gene name(symbol),第三列为分子类型(蛋白编码基因/lncRNA),第四列为节点在网络中的度。

上方红色方框中“Target Table Data”的信息表示将导入的节点属性表与之前的网络图相关联,其中“Network Collection”选择的是我们之前导入的网络文件,其他参数默认如下,可不用修改,如为其他选项,则需要通络下拉列表重新选择。
下方红色方框中“Preview”中:gene,name,molecular type,degree
第一列gene,设置为“Key”,保证gene id不重复,第二三四列均为属性“Attribute”,如需修改,同样点击名称右侧的三角形标志。
点击确定后,乍看感觉图形没有变化,但此时下方的Table Panel中已自动多出了molecular type,degree两列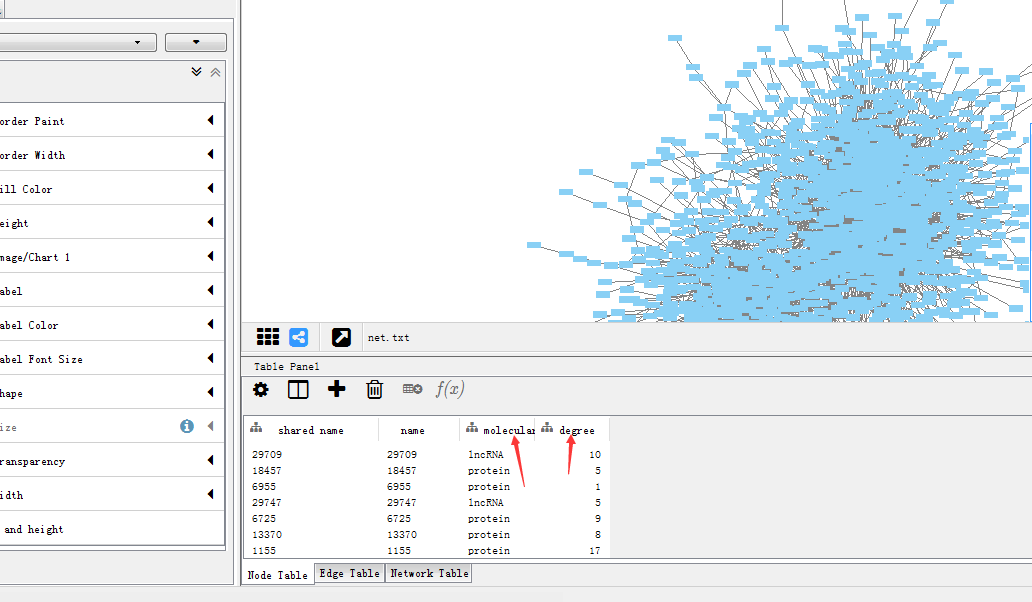
可以通过style中进行简单网络图格式设置
得到网络图:
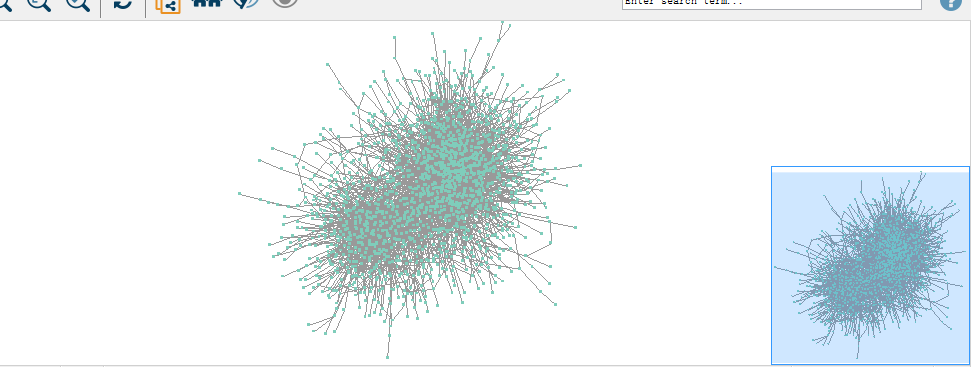
也可以自行拖拽进行微调
导出文件:数据的导出可以是网络文件,表格文件或者是图片文件,图片文件包括多种图片格式以及pdf格式,在工具栏中对应选择即可
点击菜单栏的图片导出*.pdf(同样可以采用export导出其它格式)
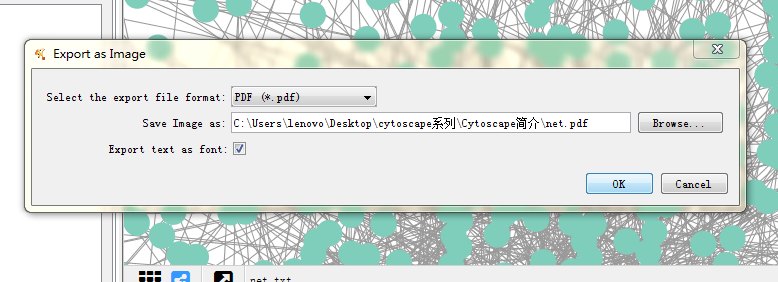
注:注意调整网络图后再保存,否则会出现网络图不完整
- 发表于 2017-09-07 12:46
- 阅读 ( 20839 )
- 分类:软件工具
你可能感兴趣的文章
- 零代码系列之三教你如何进行功能富集 4562 浏览
- 新出炉的Cytoscape视频教程 5156 浏览
- 蛋白质互作网络挖掘中Hub Genes该怎么选 23946 浏览
- 生信宝典文章集锦 5189 浏览
- String-蛋白质相互作用数据库 29653 浏览
- Cytoscape——子网提取 20394 浏览
